Kongres Akademii po Dyplomie Pediatria 2026 👉 200 zł taniej do 27.02. Spotkajmy się 17-18 kwietnia w Warszawie | Skorzystaj z oferty >
Badania naukowe
Escherichia albertii rozszyfrowana
Urszula Dąbrowska
Konsultacja medyczna: dr hab. med. Katarzyna Leszczyńska
Naukowcy z Zakładu Mikrobiologii Uniwersytetu Medycznego w Białymstoku jako pierwsi na świecie rozszyfrowali genom bakterii Escherichia albertii. Potencjalnie może ona wywoływać biegunkę
Nad rozszyfrowaniem genetycznego kodu E. albertii pracowali m.in. mikrobiolodzy z trzech amerykańskich placówek naukowych, jednak to białostockiemu zespołowi, kierowanemu przez dr hab. med. Katarzynę Leszczyńską, udało się jako pierwszemu złożyć pełną sekwencję genomu bakterii. Odkrycie to zostało zgłoszone do GenBanku, światowej bazy zawierającej zbiór genowych sekwencji nukleotydowych.
Kameleon Super
Białostoccy mikrobiolodzy przebadali materiał od ponad stu dzieci z biegunką z województwa podlaskiego. Escherichia albertii wykryto co prawda tylko w jednej próbce, ale należy ją wziąć pod uwagę jako potencjalny drobnoustrój wywołujący biegunki. Zapis genetyczny świadczy również o tym, że E. albertii to potencjalny enteropatogen, mający wiele cech wspólnych ze szczepami E. coli wywołującymi biegunki.
Do tej pory rozpoznano ponad 20 drobnoustrojów, co do których udowodniono lub wykazano, że mają potencjalny związek z biegunkami. Ciągle jednak odkrywane są nowe, dzięki zastosowaniu nowoczesnych metod diagnostycznych. Przykładem jest E. albertii, którą uznano za czynnik etiologiczny biegunek u piątki dzieci z Bangladeszu pod koniec ubiegłego wieku. Należy zaznaczyć, że wówczas zidentyfikowano ją jako Hafnia alvei, jednak szczegółowa analiza tych izolatów wykazała znaczne różnice na poziomie fenotypowym i genotypowym w porównaniu z referencyjnymi szczepami H. alvei. Na podstawie analizy 16S rRNA stwierdzono, że szczepy te największe pokrewieństwo wykazują z rodzajami Escherichia i Shigella. Ostatecznie zakwalifikowano je do rodzaju Escherichia i w 2003 roku utworzono gatunek Escherichia albertii.
W kolejnych latach pojawiały się doniesienia o izolacji E. albertii w różnych częściach świata, np. w Niemczech i w Japonii uważana jest za przyczynę kilku epidemii. E. albertii występuje również w środowisku – bakteria była izolowana m.in. od ptaków – jej rozprzestrzenianiu sprzyjać mogą ich wędrówki. Badania prowadzone w Zakładzie Mikrobiologii potwierdziły, że w naszej populacji E. albertii jest również obecna.
Należy podkreślić, że Escherichia albertii jest trudnym do diagnostyki mikroorganizmem. W standardowych metodach badawczych zachowuje się nietypowo. Komercyjne systemy do identyfikacji bakterii, stosowane powszechnie w laboratoriach mikrobiologicznych, nie rozpoznają lub błędnie klasyfikują E. albertii. Obecnie najbardziej wiarygodną metodą jej identyfikacji jest wykrycie specyficznych sekwencji DNA metodą PCR. Ponieważ szczepy tej bakterii mają gen eae, wykorzystywany jako jeden z markerów przy identyfikacji enterokrwotocznych (EHEC) i enteropatogennych E. coli (EPEC), do niedawna część z nich była błędnie identyfikowana.
– Zastosowane przez nas metody badawcze potwierdziły, że wyizolowany szczep to Escherichia albertii – mówi dr hab. med. Katarzyna Leszczyńska. – Cechy zapisane w genomie E. albertii świadczą, że może być ona potencjalnym patogenem wywołującym biegunki. Trzeba też zaznaczyć, że wśród gatunku E. albertii są zapewne szczepy o zróżnicowanej wirulencji i lekowrażliwości, co ma wpływ na ich chorobotwórczość. Jest to najczęściej powodem, że nie udaje się stwierdzić związku atypowych enteropatogenów z biegunkami. Celem naszych dalszych badań jest wykazanie, czy istnieje taka korelacja w przypadku E. albertii. Dla klinicystów jest to istotna informacja.
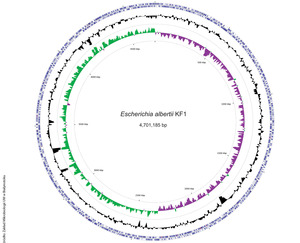
Genom E. albertii, nad którym pracował zespół naukowców w składzie: Krzysztof Fiedoruk, Tamara Daniluk, Izabela Święcicka, Emilia Murawska, Małgorzata Ściepuk i Katarzyna Leszczyńska.
Zdaniem dr Leszczyńskiej odkrycia takie potwierdzają, że w przypadku diagnostyki niektórych drobnoustrojów konieczne jest zastosowanie metod PCR, ponieważ zestawy stosowane w rutynowej diagnostyce nie zawierają w swoim spektrum np. E. albertii.
Ocena niebezpieczeństwa
Zadaniem naukowców będzie teraz przeprowadzenie na szeroką skalę badań epidemiologicznych w celu oceny częstości występowania E. albertii w różnych próbkach klinicznych i w żywności, poznania dróg rozprzestrzeniania się jej w środowisku, czynników sprzyjających kolonizacji człowieka oraz nabywania cech zjadliwości i lekooporności.
– Trudno jednoznacznie stwierdzić, czy wyizolowana E. albertii wywołała u dziecka biegunkę, ale na pewno należy wnikliwie jej się przyjrzeć jako nowemu enteropatogenowi – uważa dr Leszczyńska. – Obecnie bakterie bardzo szybko nabywają cech świadczących o ich dużej zjadliwości i lekooporności. Ma na to wpływ wiele czynników, w tym także niewłaściwie prowadzona antybiotykoterapia. Wyselekcjonowane drobnoustroje mają mechanizmy umożliwiające im szybką adaptację w niesprzyjających warunkach, zwiększenie populacji, i w rezultacie dochodzi do zaburzenia proporcji między drobnoustrojami tworzącymi mikrobiom. Przykładem jest Escherichia coli O104:H4, bakteria opisana w 2001 roku, niewywołująca wcześniej ognisk zatruć pokarmowych, sporadycznie izolowana w różnych zakątkach świata. W ciągu kilku lat wyewoluowała w hiperwirulentny szczep, który wywołał w 2011 roku epidemię w Niemczech z przypadkami śmiertelnymi.
Zespół z Zakładu Mikrobiologii UMB stara się o grant na badania epidemiologiczne, które pozwolą zbadać, jaka jest częstość występowania E. albertii w północno-wschodnim regionie Polski oraz ocenić, jaki potencjał chorobotwórczy ma ta bakteria.